1月9日,清华大学智能产业研究院(AIR)联合清华大学生命学院、清华大学化学系在《科学》杂志发表论文《深度对比学习实现基因组级别药物虚拟筛选》。该论文研发了一个AI驱动的超高通量药物虚拟筛选平台DrugCLIP, 筛选速度对比传统方法实现百万倍提升,同时在预测准确率上也取得显著突破。依托该平台,团队打通了从AlphaFold结构预测到药物发现的关键通道,首次完成了覆盖人类基因组规模的药物虚拟筛选,为后AlphaFold时代的创新药物发现带来新可能性。
长期以来,药物研发面临“高风险、高投入、低成功率”的难题,在靶点发现与先导化合物筛选阶段,受限于传统工具的计算能力,绝大多数潜在靶点和化合物仍未被充分探索。如何在广阔的生物与化学空间中精准高效地发现活性化合物,是当前创新药物研发面临的核心挑战。
据了解,为突破虚拟筛选规模瓶颈,DrugCLIP创新性地构建了蛋白口袋与小分子的“向量化结合空间”,将传统基于物理对接的筛选流程转化为高效的向量检索问题。该模型结合对比学习、3D结构预训练与多模态编码技术,能在三维结构层面精准建模蛋白-配体间的相互作用。训练后的高潜力分子将自然聚集于目标蛋白口袋的向量邻域,能够有效支撑快速的大规模虚拟筛选。依托这一机制,DrugCLIP在128核CPU+8张GPU的计算节点上,能实现毫秒级打分与万亿级日吞吐能力,筛选100万个候选分子仅需0.02秒,日处理能力达31万亿次,对比传统方法实现了百万倍提升。
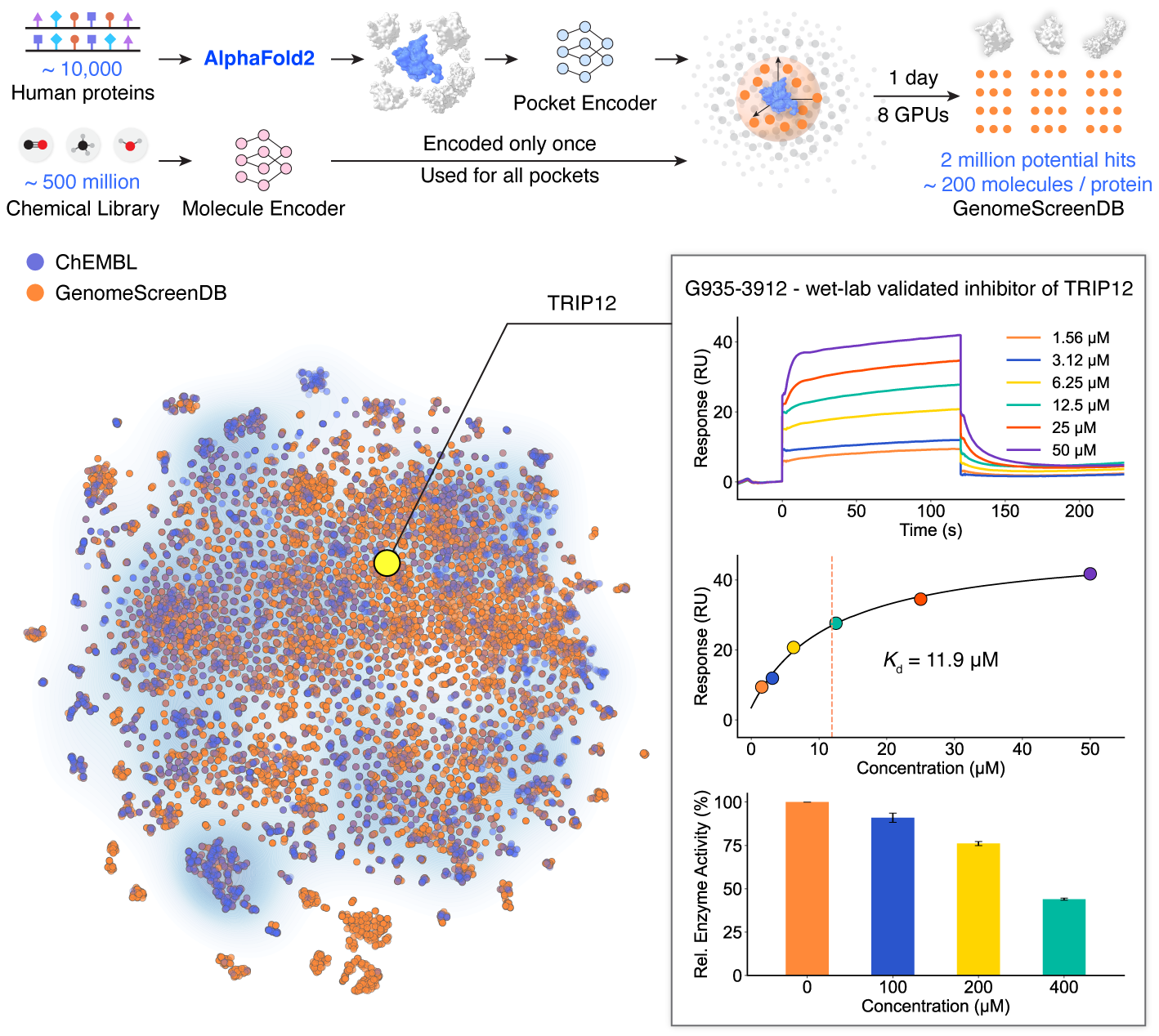
基于DrugCLIP的超高速全基因组虚拟筛选
团队与清华大学闫创业教授团队合作,在去甲肾上腺素转运体(NET)的临床相关靶点上开展了系列生物实验验证。团队使用DrugCLIP模型从160万个候选分子中筛选出约100个高评分分子,同位素配体转运实验检测显示,其中15%为有效抑制剂,其中12个分子结合能力优于现有抗抑郁药物安非他酮。相关复合物结构已通过冷冻电镜解析,进一步验证了DrugCLIP筛选结果的生物学可信度。
值得关注的是,DrugCLIP支持对AlphaFold预测的蛋白结构和apo(无配体)状态下的蛋白口袋进行筛选,扩大了其在真实药物发现场景中的适用性。团队和清华大学刘磊教授团队合作,针对E3泛素连接酶TRIP12(thyroid hormone receptor interactor 12)进行了虚拟筛选与实验验证。过往研究发现,TRIP12是多种肿瘤、帕金森综合征的潜在靶点,但是TRIP12缺少已知的小分子配体和复合物结构。团队使用DrugCLIP模型,从160万个候选分子中高通量筛选出约50个高评分分子,SPR实验证实,其中10个分子与TRIP12有结合能力,两个亲和力较高的分子也对TRIP12的泛素连接酶活性有一定的抑制活性。
此外,依托DrugCLIP,团队首次完成了人类基因组规模的虚拟筛选项目,覆盖约1万个蛋白靶点、2万个结合口袋,分析超过5亿个小分子,富集出200万余个高潜力活性分子,构建了目前已知最大规模的蛋白-配体筛选数据库。该数据库已面向全球科研社区开放,为基础研究与早期药物发现提供了强大数据支持。
DrugCLIP平台现已免费开放,用户无需本地部署,通过网页上传蛋白结构即可启动筛选任务。平台集成口袋/分子编码、向量检索、可视化与结果分析等功能,支持多种分子库调用与自定义上传,广泛适用于科研机构与企业用户。
未来,DrugCLIP将与科研产业生态合作伙伴深度合作,在抗癌、传染病、罕见病等方向加速新靶点与First-in-class药物的发现。团队将持续优化引擎性能、拓展支持模态,助力构建一个更智能、高效与普惠的全球药物创新生态。





